NGS-perusteet blogikirjoitussarjan kolmannessa osassa selvitellään auki NGS-applikaatioissa käytettyjä ja etenkin sekvensointiajoon liittyviä parametrejä ja termejä. Ensimmäinen osa keskittyy käytännön labratyön esittelyyn, toinen osa NGS-applikaatioihin ja neljännessä osassa selvitän mitä tarkoittavat UDI:t (Unique Dual indices) ja UMI:t (molecular tags/barcodes) ja miten ne eroavat toisistaan.
Sekvensointiadapterit & indeksit – UDI:t ja UMI:t, samantyyppiset sanalyhenteet, mutta mitä ne tarkoittavatkaan?
NGS-kirjastoja tehdessä olennainen osa protokollaa on sekvensointiadapterien & indeksien käyttö. Niiden tarkoituksena on mahdollistaa näytteiden identifiointi, oikeiden fragmenttien monistus ja kirjaston kiinnittyminen sekvensointilaitteiston virtauskammioon (flow cell) (nämä laitteistokohtaisia, esim. P5 ja P7 Illuminalla).
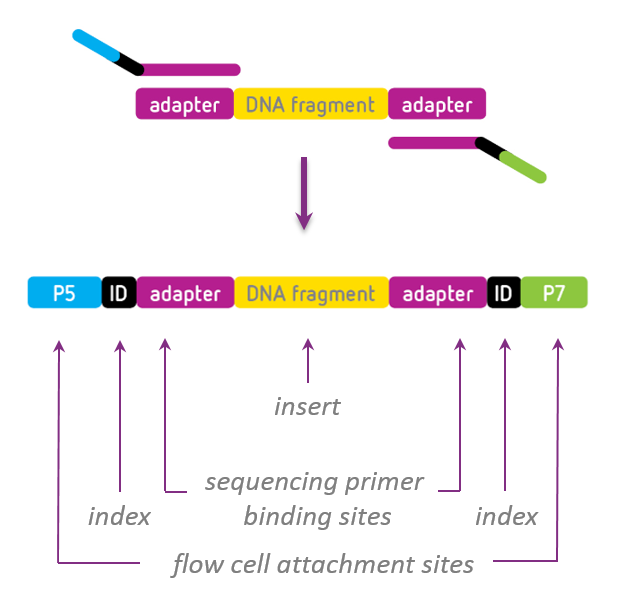
Kuva 1. Illumina indexing. Lähde https://www.gendx.com/news/35-ngsgo/illumina/245-illumina-indexing
ADAPTERIT (Sequencing adapters/adaptors):
Ensimmäinen vaihe NGS-protokollaa, ”kirjaston valmistaminen”, voi olla joko amplifikaatio eli PCR-reaktio nro 1, tai PCR-free protokollissa entsymaattinen ligaatio, jonka tarkoituksena on liittää adapterit sekvensoitavaan fragmenttiin. Tässä vaiheessa monistetaan kohde-amplikonit, tai ligoidaan adapterit esimerkiksi fragmentoituun DNA:han (jos tehdään WGS, koko genomin sekvensointia). Tässä reaktiossa käytettävissä oligoissa on mukana siis sekvensointiadapterit, ja tuloksena on kaksijuosteinen DNA/cDNA-kirjasto. Seuraavassa PCR:ssä nro 2, (”indeksointi-PCR”), monistetaan edellisessä vaiheessa valmistettu kirjasto. Nämä PCR-primerit tunnistavat edellisessä stepissä käytetyt sekvensointiadapterit (indexed adapter oligos). Tämän PCR:n jälkeen sinulla on ”viivakoodatut” näytteet valmiina sekvensoitavaksi.
INDEKSIT
Käytetään aina sekvensoitavien näytteiden identifioimiseksi, ja indeksit mahdollistavat näytteiden multiplexauksen. Indeksit ovat noin 6-10 emäksen pituisia lyhyitä oligoja jotka liitetään tutkittavan fragmentin päihin.
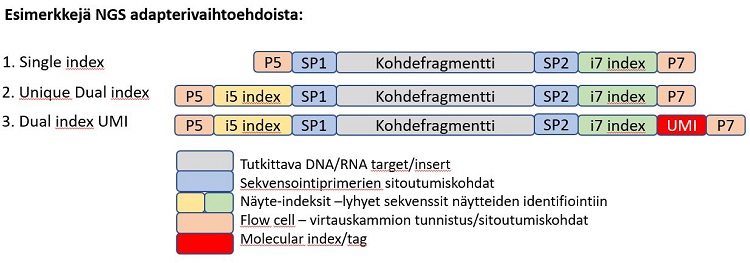
Single-index: Single indeksejä käytettäessä mukana on aina i7 adapteri ja näytteitä voidaan poolata yhteen ja sekvensoida esim. 96 kpl kerralla yhdessä ajossa.
Dual Indices: Käytettäessä ”tavallisia”, Combinatorial kaksoisindeksejä, ne liitetään PCR nro 2:ssa fragmentin molempiin päihin. Esim. käytettäessä neljää (4) eri indeksiä, mahdollistetaan 6 eri indeksikombinaatiota näytteiden välille, joista 4kpl on uniikkeja. Kaksoisindeksit ovat nimeltään i7 (Index Read 1) ja i5 (Index read 2).
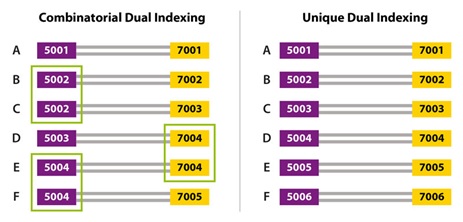
Kuva 2. Vasen; tavallinen indeksointi; jokaisella näytteellä/kirjastolla A-F on oma yhdistelmä i5-i7 indeksejä, mutta joitakin indeksejä on voitu käyttää useamman eri näytteen kohdalla (5002, 7004). Oikealla, esimerkki UDI-kirjastoista, joissa jokaista indeksiä käytetään ainoastaan kerran. (www.lexogen.com)
Unique dual indices UDI:t käytettäessä, fragmentin molempiin päihin liitetään indeksit, joiden sekvenssit ovat erilaiset. UDI:jä käytettäessä voidaan multiplexata satoja näytteitä yhteen ajoon. Täten kuhunkin näytteeseen (eri potilaat, eri näytteenottokohdat, eri aikapisteet) saadaan uniikki ”viivakooditunniste” aikaiseksi.
Käytännössä tämä tapahtuu siten, että PCR:ssä nro 2 käytetään primereitä eli oligoita, joissa on kaikki nämä komponentit: adapterin tunnistussekvenssi, indeksi (6-10 nukleotidiä) ja P5 tai P7 sekvenssipätkät jotka sitoutuvat virtauskammion (flow cell) pintaan.
Sekvensoinnin yhteydessä, laitteistolle kerrotaan mikä indeksi liittyi mihinkin näytteeseen (sample sheet excel-taulukko). Sekvenssiajon jälkeen, data-analyysiohjelmat pystyvät erottelemaan indeksit ja täten eri näytteet toisistaan, ja oikeat readit pystytään yhdistämään oikeaan näytteeseen (de-multiplexing of data).
Unique Dual Indeksien hyödyt:
- Mahdollistaa näytteiden tehokkaan ja tarkan multiplexauksen
- Mahdollistetaan tarkempi ja luotettavampi data, esim. harvinaisemmat variantit pystytään tunnistamaan paremmin (etenkin low input-näytteiden osalta, esim. mutaatioanalyysi soluvapaasta DNA:sta, tai harvinaisempien transkriptien detektio RNA-seqissä)
- Tunnistaa ja määrittää index hopping (ilmiö Illuminan NovaSeq-sekvensoinnissa) ja välttää indeksien väärää tunnistusta (=väärä sekvenssidata väärästä näytteestä; avoid cross-contamination)
Käytäntö labrassa: UDI:jä tarjoaa vain muutama valmistaja, ja ne on yleensä valmiina 96-levyillä. 96 erilaista indeksiä mahdollistaa siis 96 uniikkia kombinaatiota ja 9216 (96 x 96) erilaista kombinaatiota. Indeksit tulevat joko helposti kirjastokittien mukana (”premixed” kirjastonvalmistusreagenssien kanssa) tai erillisenä tuotteena, ja joskus ne tarvitsee tilata erikseen toiselta toimittajalta.
Meiltä UDI:t saatavilla Lexogenin RNAseq-tuotteisiin, NuGENin DNA- ja RNAseq-kirjastokittien mukana, ja Swift Biosciences DNA-kirjastokittien mukana.
 UMI: Unique Molecular Identifiers tai Unique Molecular Tags/barcodes
UMI: Unique Molecular Identifiers tai Unique Molecular Tags/barcodes
UMI:en eli molecular tagien tarkoituksena on tunnistaa mahdollinen PCR-bias kirjastonvalmistuksen aikana.
NGS-protokollissa on PCR-reaktioita, joiden aikana voi tapahtua epäspesifistä sitoutumista ja monistua vääriä fragmentteja, jotka myös päätyvät sekvensoitavaksi. Tätä tapahtuu etenkin pieniä low input -näytemääriä käytettäessä (esim. single cells, cell-free DNA). Esimerkiksi RNAseqissä, kahden eri geenin ja transkriptin cDNA-saannot voivat olla yhteneväiset, mutta PCR:ssä toisia cDNA-fragmentteja monistetaan mahdollisesti tehokkaammin kuin toisia ja tämä vääristää todellista ekspressiotasojen kvantitointia.
UMI:t ovat lyhyitä emäspätkiä, random 6-10 nt oligoja jotka ligoidaan DNA-fragmentteihin ennen PCR:ää. Määrittämällä UMIen määrä PCR:n jälkeen, saadaan tarkempi kuva todellisesta fragmenttien kopiomäärästä per geeni/transkripti, ja PCR-duplikaatit voidaan tunnistaa. UMI-sekvenssi on usein osa indeksi-readia ja osana kirjastokittiä, mutta ne voidaan tilata myös erikseen. Eri toimittajien tarjoamat UMI:t eroavat lähinnä sen suhteen missä osassa adapteria ne sijaitsevat (voi olla esim. osa index readia i5).
Jos kaipaat molekulaaristen tagien käyttöä RNA-seqiin, meiltä ne on saatavilla Lexogenin QuantSeq-ekspressioprofilointiin sekä esim. single cell-näytteiden koko transkriptomianalyysiin soveltuvan NuGEN:in kirjastokitin mukana.
Lisäksi, Unique Molecular identifiers UMIs hyödynnetään Paragon Genomicsin superherkässä Lung cancer paneelissa. Jopa 0.1% alleelifrekvenssin detektioherkkyys; soveltuu cell free DNA:n /nestebiopsiat tai circulating tumor cells ctDNA:n analysointiin, myös syöpäprofilointi FFPE-näytteistä.
Tutustu myös NGS-perusteet blogikirjoitussarjan muihin kirjoituksiin.
Blogin kirjoitti Sanna Siltanen, PhD.
