Valikoimissamme useita vaihtoehtoja koronaviruksen NGS-sekvensointiin; amplikonipaneelit joko cDNA-käännön kanssa tai ilman sekä erilaiset RNA-sekvensointiratkaisut/kirjastokitit, lue lisää! Tutustu myös helppoon ja nopeaan NGS Data-analyysiratkaisuun!
Swift Biosciences SARS-CoV-2 Additional Genome Coverage amplicon panel
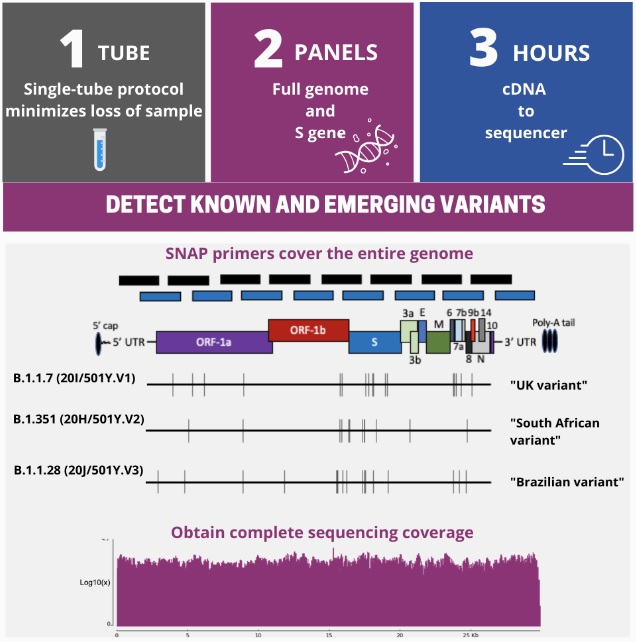
- Utilize multiple overlapping amplicons in a single tube
- Rapid, 2-hour PCR1+PCR2 workflow generates robust libraries even from low input quantities
- produces libraries from first- or second-strand cDNA produced from viral isolates or clinical specimen.
- This unique design enables successful SARS-CoV-2 library preparation from samples with low viral titers and provides powerful solutions for the confident detection of nucleotide variants.
- Reagents included: target-specific multiplex primer pool, PCR and library prep reagents, Swift Normalase reagents, combinatorial dual indices and up to 1536plex UDIs
- Swift protocol 2- 3hours cDNA-to-Library: PCR1 single pool (1h 10min), 1st mag bead cleanup, indexing-PCR2 (35min), 2nd cleanup + QC (+optional downstream Normalase step to produce an equal molar library pool.)
- Sequencing: 50K reads/library (+/-) detection; 1M reads per library (variant calling)
UUTUUS! Swift Biosciences S GENE panel –
The SPIKE (S Gene) covered as a separate panel
- SARS-CoV-2 genomics coordinates covered: 21497-25421
- Provides 100% coverage of the S Gene from limited viral titres
- Perform multiplex PCR in a single tube reaction
- Robust yields from low input samples using PCR1+PCR2 workflow
- Supports high-throughput library quantification methods (fluorometric, electrophoretic, or Swift Normalase)
- Designed for targeted pathogen sequencing to enable variant detection
- Optimized for all Illumina® sequencing platforms
Paragon Genomics CleanPlex® SARS-CoV-2 FLEX Panel
- Designed for Coronavirus detection, tracking, research and surveillance, enabling complete genome sequencing of the new SARS-CoV-2 virus.
- The FLEX panel is built upon the original SARS-CoV-2 Panel
- Contains additional components for more robust and confident variant calling even when the virus mutates over time.
- Differs from the standard SARS-COV-2 Panel in that it contains degenerate primer designs of polymorphic regions of the genome to allow more robust amplification of variable strains
- The FLEX panel also contains a human positive control that amplifies at 100bp longer than the viral library peak to serve as an internal library preparation control, especially for negative samples.
- The entire genome of the virus can be amplified from RNA to sequence-ready libraries in 5.5 hours
- Reagents included: RT module, PCR primers, lib prep reagents, plated UDIs, mg purification beads.
- Sequencing: Only 0.2M reads per sample (with 2x 150 PE)
- Sample multiplexing: MiSeq® System (v2 chemistry Nano): 39 samples, MiSeq System (v2 chemistry): 583 samples, MiniSeq™ System (mid-output): 311 samples, MiniSeq System (high-output): 972 samples, With the multiple flow cell capability on a NovaSeq™ 6000, one can sequence up to 3,072 samples simultaneously for faster turn around and higher throughput workflows.
- Paragon FLEX protocol 5-6hour (1h hands-on): RT reaction = cDNA synthesis, target amplification (2 pools), background cleaning, indexing & amplification
Paragon Genomics CleanPlex® SARS-CoV-2 NGS Paneeli (RUO)
- Multiplex-PCR -pohjainen amplikonipaneeli
- Kattaa koko virusgenomin (lukuun ottamatta 92 bp lopusta)
- Target size: 29 930 bp
- 343 amplikonia (kahdessa eri primer-poolissa)
- input 50 ng total RNA
- High Coverage – 99% coverage of the entire SARS-CoV-2 genome
- Herkkä- tunnistaa 1 viruskopion (vs. 3-5 kopiota RT-qPCR-testillä)
- Workflow:n kesto 5.5h (reverse transcription, multiplex-PCR, CleanPlex taustankorjaus, indeksointi- PCR, poolaus) + sekvensointi, hands-on aika 1h
- Soveltuvuus Illumina & MGI laitteistoille
Paragon Genomics CleanPlex® Respiratory Research Panel v2
Designed for COVID-19 Influenza (flu A/B), and RSV (A/B) detection, research, and surveillance, enabling complete genome sequencing of the SARS-CoV-2 virus and concurrent influenza A and B sub-typing with highly sensitive detection. The respiratory virus primers were expertly designed in consideration of over 45,000 viral genome sequences to generate highly specific, but also universally conserved targets for each influenza and RSV subtype to allow confident detection throughout flu seasons, regardless of changes in strain prevalence. Ready for early access customers. Lue lisää
Paragon Genomics CleanPlex® ACE2 & TMPRSS2 Panel
A multiplex PCR-based targeted sequencing assay designed to examine two genes associated with COVID-19 susceptibility. Interrogates entire ACE2 & TMPRSS2 coding and promotor regions plus curated 70+ eQTL targets. Lue lisää
Webinar: Turn your sequencing lab into a COVID-19 research and surveillance powerhouse
Swift Biosciences SARS-CoV-2 PANEL (RUO)
- koko virusgenomin (98%) kattava amplikonipaneeli
- input: 1ng virus-cDNA:ta
- Helppo & suoraviivainen protokolla: 2h:ssa cDNA:sta kirjastoksi
- Laitesoveltuvuus: Illumina-sekvensointi
- Näytteiden multiplexaus: 96 / 384 CDI per MiSeq ajo
- Kittikoko: 48rxn, sis. PCR primerit, kirjastonvalmistusreagnessit, indeksit
Swift Amplicon SARS-CoV-2 -paneeli on robusti menetelmä, jolla saadaan aikaan optimaalinen lukupeitto. Perustuu patentoituun multiplex-PCR teknologiaan, joka mahdollistaa kirjaston rakentamisen 1. juosteen tai toisen juosteen cDNA:sta käyttämällä rinnastettuja alukepareja. Alukkeet on suunniteltu NCBI:n referenssisekvenssin NC_045512.2 mukaan. In Silico-analyysissä ei tunnistettu vastaavia kohdesekvenssejä ihmisen isäntägenomisekvensseistä. Lue lisää
ACE2-geenipaneeli – ACE2 SWIFT NORMALASE® AMPLICON PANEL (SNAP)
- Catalog No ACE2G1-96, Swift Biosciences
- Multiplex-PCR-pohjainen NGS-amplikonipaneeli, workflow 2.5h
Ihmisen hengitysteiden epiteelisolujen pinnalla olevan ACE2: n on todettu olevan avainreseptori SARS-CoV-2:n pääsyyn isäntäsoluihin. Vaikka SARS-CoV käyttää myös ACE2: ta reseptorina, SARS-CoV-2 viruksen mutaatiot lisäävät sen sitoutumisaffiniteettia ACE2: een suhteessa SARS-CoV: hen. ACE2-reseptorin sekvenssin ja ekspressiotasojen vaihtelut voivat vaikuttaa SARS-CoV-2: n kykyyn sitoutua onnistuneesti ja siirtyä isäntäsoluihin, ja lukuisat ACE2-variantit onkin liitetty sairauden alttiuteen ja vakavuuteen. ACE2: n sekvensointi ja geenin varianttien tutkiminen voi antaa lisätietoa COVID-19 infektion vakavuudesta ja helpottaa epidemiologisia lisätutkimuksia. Lue lisää
Swift Normalase® Amplicon SARS CoV-2 Panel (SNAP)
SNAP kits utilize multiple overlapping amplicons in a single tube, using a rapid, 2-hour workflow to prepare ready-to-sequence libraries. The PCR1+PCR2 workflow generates robust libraries, even from low viral load samples. The libraries may be quantified with conventional methods such as Qubit® or Agilent Bioanalyzer and normalized by manual pooling or normalized enzymatically with the included Swift Normalase reagents.
Uusin julkaisu: Sensitive recovery of complete SARS-CoV-2 genomes from clinical samples using Swift Biosciences’ SARS-CoV-2 multiplex amplicon sequencing panel. Addetia A et al., J Clin Microbiol Oct 2020
“In summary, the Swift SARS-CoV-2 amplicon panel is a simple, highly sensitive approach for recovering SARS-CoV-2 genomes. The panel has allowed for the study of genomic rearrangements and mutations that are uniquely associated with low viral load samples.”
Lue lisää Swift Biosciences COVID-19 NGS-ratkaisujen valintaoppaasta!
Lexogenin RNAseq-ratkaisut
Lexogenin RNAseq-ratkaisut/transkriptomiikka: virus-RNA:n sekvensointi, ribosomaalisen RNA:n depleetion kanssa (poistetaan humaaninäytteistä human rRNA) mahdollistaa virus-RNA:n rikastuksen ja täten kustannustehokkaan sekvensoinnin. Saatavilla myös cDNA-kääntökitti TeloPrime sekä RNAseq-kirjastokitti QuantSeq-Flex, johon mahdollista sisällyttää mitkä tahansa custom-primerit viruksen kohdegeenien sekvensoimiseen (gene expression panel). Myös helppokäyttöinen RNA-sekvenssin data-analyysiratkaisu saatavilla. Lue koko informatiivinen kooste täältä
NuGEN/Tecan Genomics Trio-RNAseq
- RNAseq ratkaisu koronaviruksen sekvensointiin ja identifikaatioon
- soveltuu low input -näytteille (alkaen 500 pg total RNA)
- sisältää myös humaani-rRNA:n depleetioprobet, mikä parantaa herkkyyttä ja mahdollistaa tarkemman virusdetektion. Lue lisää
”The advantage in using Trio RNA-Seq is that it effectively enriches for viral load through utilising Tecan Genomics single primer isothermal amplification (SPIA) + AnyDeplete for the removal of non-informative rRNA transcripts. This effectively increases the dynamic range of detection but also improves the quality of the sequence data. “
Referenssit, kaksi tuoretta julkaisua:
Genomic diversity of SARS-CoV-2 in Coronavirus Disease 2019 patients, Shen Z et al.
RNA based mNGS approach identifies a novel human coronavirus from two individual pneumonia cases in 2019 Wuhan outbreak, Chen L et al.
NuGEN/Tecan Genomics Revelo
NuGEN/Tecan Genomics Revelo (pohjautuu Trio-RNAseq -kittiin jossa SPIA, single primer isothermal amplification teknologia):
- High detection sensitivity of SARS-CoV-2
- Detect down to ~500 viral copies with minimal sequencing read depth
- Identify co-infective agents in the same sample, without a priori knowledge
- Input: 250 pg – 10 ng total RNA
- Lib prep kit specifically designed to deal with the problems of detecting and amplifying viral pathogens from degraded samples
- Simple, rapid, user-friendly protocol that can take you from sample to sequencer in just ~6.5 hours, lataa tästä esite